单细胞测序之条形码水凝胶微球 --Barcoded Hydrogel Microspheres (一)
2025-02-10
2025-02-10
2015年来自哈佛医学院的Macosko等人建立了一种Drop-Seq技术平台,用于高通量快速分析小鼠视网膜细胞的转录组,创新性地引入了barcoded beads (条形码微球),进行mRNA的测序。原理如下(图1):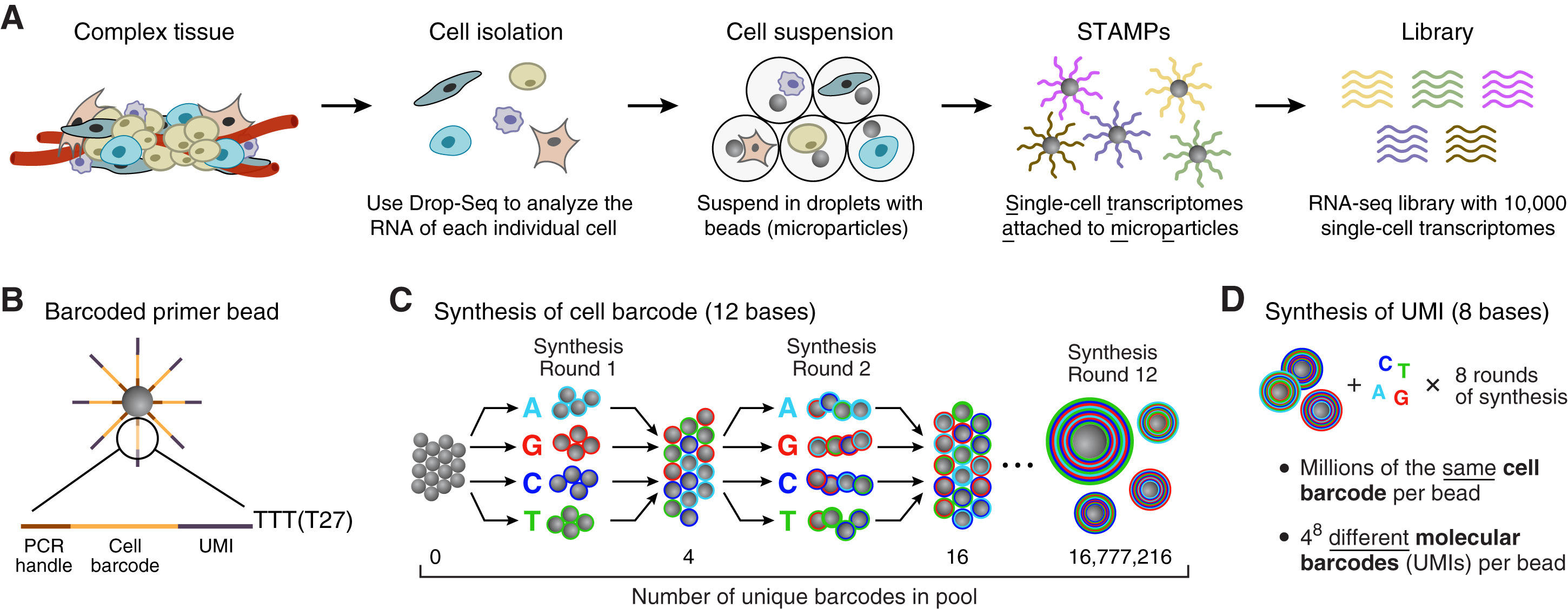
A为分析流程,(1)从组织中制备单细胞悬浮液;(2)将每个细胞与带有不同条形码的微粒(珠子)共同封装在纳升级的液滴中;(3)将细胞分离到液滴中后进行裂解;(4)通过条形码序列末端的poly dT捕获 mRNA,形成 STAMP(附着在微粒上的单细胞转录组);(5)在一次反应中逆转录、扩增和测序数千个 STAMP;(6)使用 STAMP 条形码推断每个转录本的来源细胞。B-D为条形码微球的制备,在此采用的是split-pool(分裂池法)。分裂池法是一种是指数形式生成产物的高效合成技术,在这儿,采用的是单个碱基添加的办法,有4种碱基,所以分为4个分裂组,在后面我们会采用短核苷酸链,从而可以分为更多的分裂组(如96次,刚好一个96孔板),减少编码的轮次。具体的过程,感兴趣的可以私下讨论。在编码完成后,后面再添加UMIs(Unique Molecular Identifiers)有捕捉mRNA的poly -T。图B为编码球的序列结构,依次(1)恒定序列(PCR Handle,在所有引物和珠子上相同),用作下游 PCR 和测序的引发位点;(2)细胞条形码(Cell barcode,;(3)唯一分子标识符 (UMI),每个引物上不同,以识别 PCR 重复;(4)寡 dT 序列,用于捕获多聚腺苷酸化的 mRNA 并引发逆转录。
Indrop-Seq (indexing droplets)也是哈佛医学院在2015年建立的,主要由Klein等人完成。当时是用于高通量快速分析小鼠胚胎干细胞的mRNA,原理如下(图2):
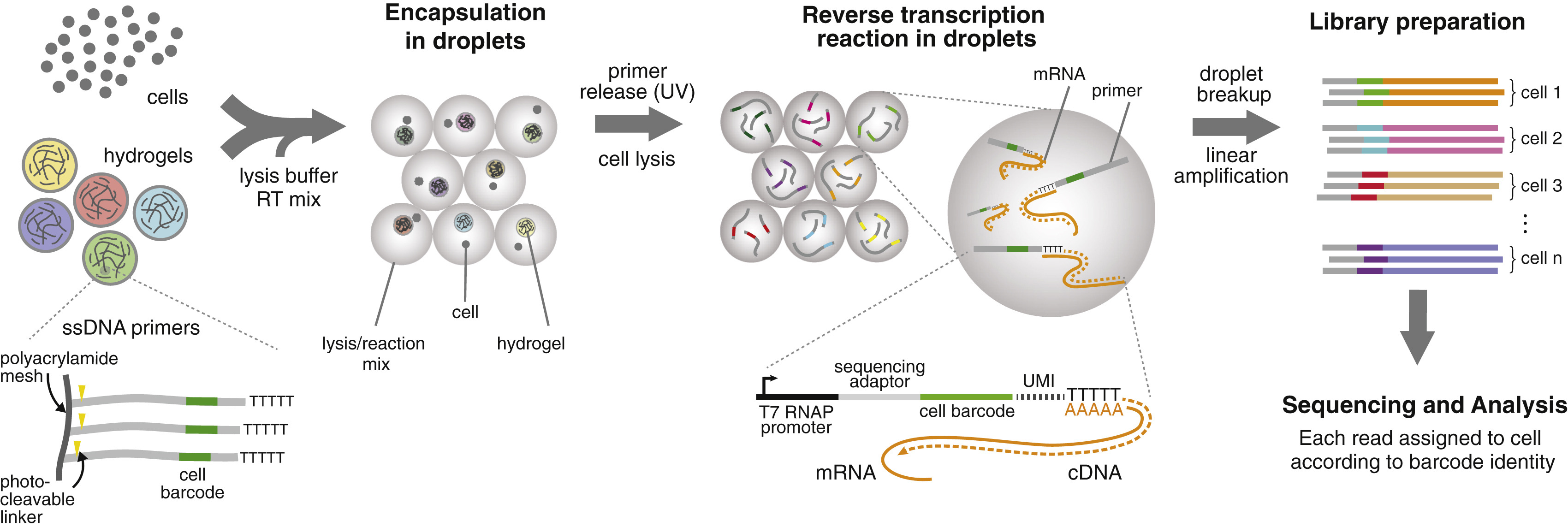
图2. inDrop-seq 分析流程
inDrop条形码水凝胶微球的制备流程。微球通过丙烯酸亚磷酰胺与光敏spacer ispPC结合,后面为T7 启动子序列,测序序列(PE1),形成一个所谓的通用DNA引物进入第一轮次的编码。第一轮次编码序列的结构为PE1*+barcode1*+W1*, 通用的DNA引物可以据此为模板,进行扩增得到“通用DNA引物+barcode1+W1”,混合再分组后,进入第二轮次的编码,第二轮次用的序列结构为W1*+Barcode2*+UMI+poly-A, 以DNA引物+barcode1+W1上的W1为引物,以第二轮次用的序列结构为模板进行扩增得到"通用DNA引物+barcode1+W1+bacode2+UMI+poly-T的微球条形码序列(每轮次的chemical blocks为384,因此条形码数理论为384x384=147456)。详细见图3:
图3. inDrop条形码微球制备
HyDrop-seq 是鲁混汶大学的Florian V De Rop 等人所建立,他们设计了两种水凝胶编码球 分别用于scATAC-Seq scRNA-Seq, 前者用于单细胞ATAC分析(染色质开放区域DNA测序),后者是mRNA测序分析。相对于inDrop-Seq 和Drop-Seq,其灵敏度及数据产出方面均有极大的提升。Hydrop 条形码微球的制备与inDrop条形码微球相似,下面是其流程(图4):
图4. Hydrop条形码微球的制备Florian V De RopJoy N IsmailCarmen Bravo González-BlasGert J HulselmansChristopher Campbell FlerinJasper JanssensKoen TheunisValerie M ChristiaensJasper WoutersGabriele MarcassaJoris de WitSuresh Poovathingal Stein Aerts (2022) Hydrop enables droplet-based single-cell ATAC-seq and single-cell RNA-seq using dissolvable hydrogel beads eLife 11:e73971.
Klein AM, Mazutis L, Akartuna I, Tallapragada N, Veres A, Li V, Peshkin L, Weitz DA, Kirschner MW. 2015. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell 161:1187–1201. DOI: https://doi.org/10.1016/j.cell.2015.04.044, PMID: 26000487
Macosko EZ, Basu A, Satija R, Nemesh J, Shekhar K, Goldman M, Tirosh I, Bialas AR, Kamitaki N, Martersteck EM, Trombetta JJ, Weitz DA, Sanes JR, Shalek AK, Regev A, McCarroll SA. 2015. Highly Parallel Genome-wide Expression Profiling of Individual Cells Using Nanoliter Droplets. Cell 161:1202–1214. DOI: https://doi.org/10.1016/j.cell.2015.05.002, PMID: 26000488
上一篇
DSS 小鼠结肠炎造模下一篇
无